定期通信第41号は、当法人の豊福 肇 理事による書下ろし「微生物検査データの標記と解釈」です。
是非、ご覧ください。
微生物検査データの標記と解釈
豊福 肇 (山口大学 共同獣医学部 病態制御学講座 教授)
平成30年6月の食品衛生法等の一部改正によりHACCPが制度化された。今後、微生物検査は従来の最終製品の出荷の可否を判定する検査から、妥当性確認及び検証のツールとして、役割が変わるが、重要性は増加すると考えられる。 本稿においては、そのかなり前段として、微生物の検査において、どのように菌数を標記し、解釈するか、その時の注意点について紹介する。
菌数はlog(常用対数)に変換して標記され、解釈される。これは、細菌は2,4,8,16と二分裂で増えていくため、条件が整えば、短時間で対数的に増加し、“109/g”にまで増加しうる。そこで、解釈をしやすくするために対数変換を行う。また、減少についても“2 log reduction/g”のように標記され、これは菌数が1/100になる=99%の菌が死滅することを意味する。
“2 log reduction”は“6 log CFU/g”から“4 log CFU/g”あるいは“4 log CFU/g”から“2 log
CFU/g”など、どの菌数からスタートしても“2 log reduction”は同じである。“3 log reduction”は99.9%減少、“4 log
reduction”は99.99%減少となる。缶詰のボツリヌス菌の“12 log reduction”は、すなわち、99.9999999999 %減少を意味する。
このlog 変換は計算する時に注意が必要である。例えば、10,000 CFU/g (4 log10 CFU/g)の濃度の菌を含む食品10gと、同じ菌を1,000 CFU/g (3log10
CFU/g)の濃度で含む食品10gがあるとする。このそれぞれの食品には100,000 CFU 及び 10,000 CFUの菌が存在することになる。この食品を混合すると、食品20g中に合計110,000 CFU (1.1×105
CFU)の菌 が存在し、濃度は110,000/20g = 5,500/g= 5.5×103 CFU/g (すなわち 3.74 log10 CFU/g)となる。
ここで良く犯す間違いは2つの“log10濃度”(4 log10 CFU/g及び3 log10CFU/g)を加算して、混合した食品に“7 log10 CFU/g”(すなわち10,000,000 CFU/g)としたり、また、2つの“ log10濃度”の平均(すなわち3 log10 CFU/g及び4 log10 CFU/g) をとって、“3.5 log10CFU/g”とするものである。“3.5 log 10 CFU/g”は3,162 CFU/gなので、真の平均である5,500 CFU/gより低く推定されてしまう。
通常はlog10の数値で菌数をあらわすが、リスク評価の場合等、算術スケールに変換する必要が生じる。食品中の菌は対数正規分布で分布すると考えられている。例えば、平均が“3 log10 CFU/g”で、標準偏差が“0.8 log10 CFU/g”の分布を考えた場合、この分布を算術スケールに変換すると、算術平均は“5,455”、幾何平均は“1,000”、標準偏差は“29,257”となる。
幾何平均“3log10” =1,000と算術平均が5倍違うことに注意が必要である。ここで、標準偏差を“0.2 log10
CFU/g”に変えた場合、算術平均は“1,112”、標準偏差は“540”と変化する。標準偏差を“0.4 log
10
CFU/g”に変えた場合、算術平均は“1,528”、標準偏差は“1,766”となり、標準偏差を“0.6 log10
CFU/g”に変えた場合、算術平均は“2,597”、標準偏差は“6,224”と変化し、標準偏差を“1.2 log
10
CFU/g”とした場合、算術平均は“45,484”、標準偏差は“2,068,319”と変化する。この変換を正しく行わないと、誤解することになる。
上記の計算はWHOの以下のサイトからエクセルシートをダウンロードして自ら計算することができる。
https://www.who.int/foodsafety/publications/mc-tool.xls/en/
また、その使用法は、
https://www.youtube.com/watch?v=iQWCnykNKWQ&feature=youtu.be
で見ることができる。(英語)
このように、微生物の分布においては標準偏差が極めて重要になってくる。食品中の菌の標準偏差のデータがない場合は、
- 液体食品のように、よく混和されている食品の場合には、0.2 log10 CFU/g
- ある程度混和されている食品の場合には、0.4 log10 CFU/g
- あまり均一ではない食品の場合には、0.8 log10 CFU/g
を仮定している。
リステリアのサンプリングプラン
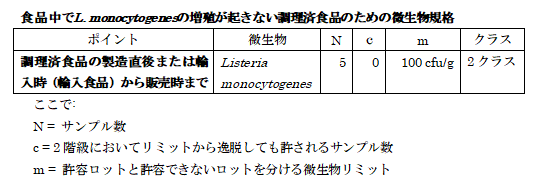
この微生物規格はISO11290-2 法またはISO16140に基づく適切な感受性、再現性及び信頼性に関する同等性評価を受けた他の方法を用いることをベースにしている。
対数正規分布を仮定し、このサンプリングプランでは、5サンプル中1検体でも100 CFU/g
L. monocytogenesを超えることにより、幾何平均濃度93.3 CFU/g、分析の標準偏差が0.25 log10 CFU/gのL. monocytogenesを含む食品ロットを、95%の信頼性で検出し、拒否できる。そのようなロットは、55%のサンプルは100 CFU/g未満、 45 %までのサンプルは100 CFU/gを超えていて, 全サンプル中0.002 %は1,000
CFU/gのレベルの菌で汚染されている。
このように、コーデックスの微生物規格では1ロットから採取すべきサンプル数、リミット、リミットを超えることが許容されるサンプルの数、検査法及び検査法のパフォーマンスが規定される。サンプル数Nを増やせば、またcを小さくすれば、そのサンプリング計画の厳しさは増す(=より安全な食品になる)。このサンプリング法を用いて合格したロットであっても、その45%のサンプルは100CFU/gを超えるL. monocytogenesに汚染されていることがあり得る。
この割合を下げたければ、Nを増やす(例えばN=10にした場合には、100CFU/gを超えるL. monocytogenesに汚染されている可能性は25.9%に、N=20にした場合にはそれが13.9%にまで下がる)ことが必要となる。
参考文献
WHO. Statistical Aspects of Microbiological Criteria Related to Foods
MICROBIOLOGICAL RISK ASSESSMENT SERIES 24, A RISK MANAGERS GUIDE
https://www.who.int/foodsafety/publications/mra_24/en/
Codex Alimentarius. Guidelines on the application of general principles of food hygiene to the control of Listeria monocytogenes in foods (CAC/GL 61-2007)
以上
